Discovery
This branch of research investigates the highly complex interactions of different immune cells—with each other and with the organism’s cells—to understand how the immune system maintains the health of the organism.
We develop innovative therapies for the treatment of cancer, autoimmunity, and chronic inflammation. By reprogramming immune cells through synthetic and pharmacologic intervention, we build cells that save lives.
Our scientific activities are structured into three Research Areas: Discovery, Translation, and Clinical Application. All three work in synergy with one another. Our work starts with basic research into the areas of immune regulation, immune metabolism, cancer, and tissue homeostasis. It carries on with a focus on therapy development—spanning the creation of new formats of genetic and pharmacologic cell manipulation and drug-compliant manufacturing processes. Finally, we seek to apply our discoveries in early clinical trials on patients themselves.
This branch of research investigates the highly complex interactions of different immune cells—with each other and with the organism’s cells—to understand how the immune system maintains the health of the organism.
This area focuses specifically on the preclinical development of immune-cell therapeutics. This includes the identification, differentiation, expansion, and preclinical testing of therapeutically relevant immune cell populations.
At the LIT, our core focus is on the clinical translation of scientific findings: We therefore place significant emphasis on the development and roll out of clinical trials and focus on the clinical implementation of the results.
Together we stand to find new solutions for the treatment of diseases with unmet need.
View All PIs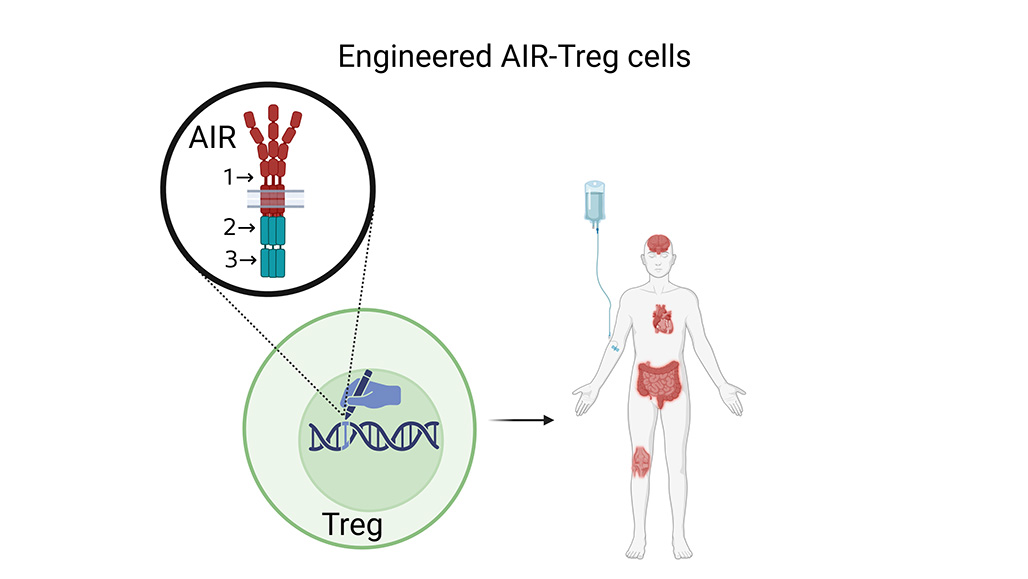
Scientists from the Division of Immunology have developed new types of synthetic biosensors known as artificial immune receptors (AIRs), which can be used to reprogram Tregs. These AIR receptors equip Tregs with a novel inflammation-sensoring mechanism and translate this environmental information into a dependent TCR-activation program. Learn More
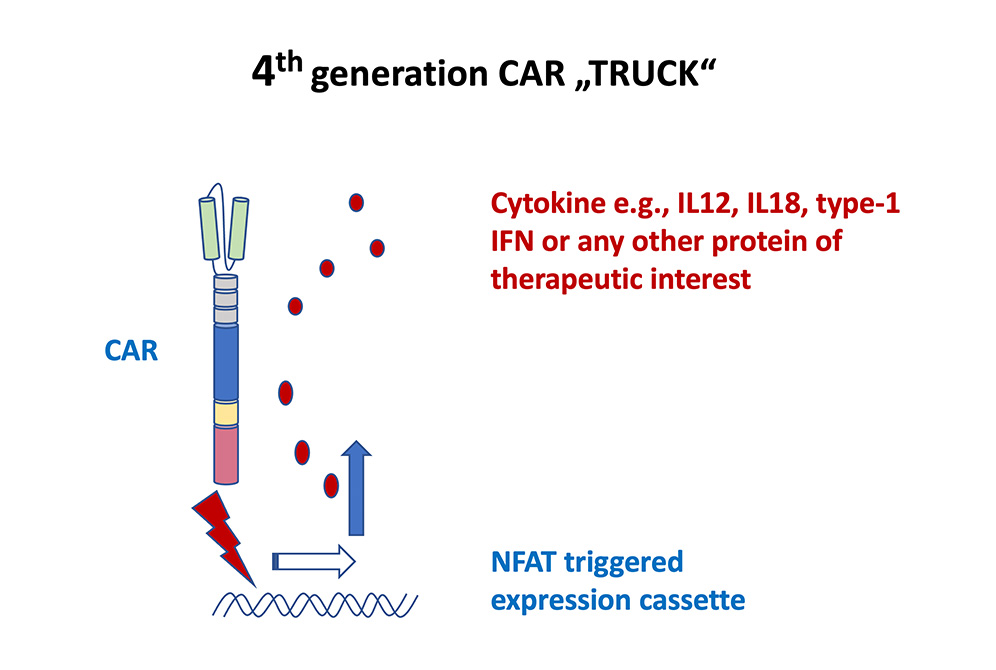
The Leibniz Institute aims to use CAR T cells not simply as ‘living drugs’ in themselves, but also as ‘living factories’ producing a transgenic protein which delivers a therapeutically active agent to the organ of interest. The T cells are engineered with a CAR and an inducible expression cassette to produce a therapeutic protein which is triggered by CAR signaling. Learn More
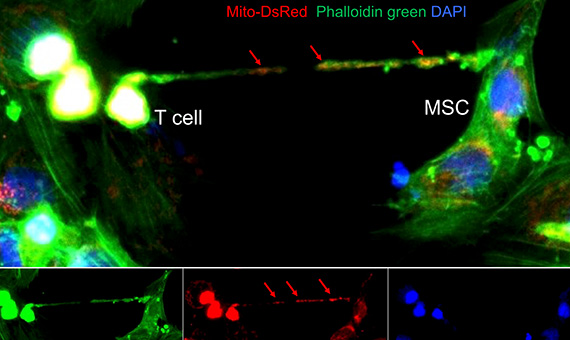
Immunological memory describes the immune system’s ability to remember a pathogen, and respond rapidly and more vigorously to it upon subsequent encounters. This essential immune system function is delivered by B and T cells. Mesenchymal stromal cells (MSCs) in the bone marrow and peripheral lymphoid organs are known to promote T-cell memory by organizing pro-survival niches—but the precise mechanisms by which they support T-cell longevity are still not clearly understood. Learn More










